你在这里
2007年4月期刊俱乐部主题:生物分子的分析建模
 a的分子组成部分细胞包括:
a的分子组成部分细胞包括:
♦膜组件(如。脂肪酸而且磷脂)
♦生物聚合物(如。蛋白质)
基因蓝图(如:DNA而且核糖核酸)
在一个俱乐部杂志的前一期,约翰Dolbow讨论了生物膜的计算力学。我想从分析的角度来讨论蛋白质和DNA (RNA)的力学问题。
蛋白质是蛋白质的线性链氨基酸它们是主成分细胞骨架也存在于大多数膜中。蛋白质是细胞的重要功能元素,在许多情况下,它们的结构或机械功能与它们的生化作用一样重要。细胞骨架中的蛋白质可以形成一个支架系统,以维持细胞的完整性;此外,许多基本重要的生物过程依赖于蛋白质及其组合的机械反应,包括转导而且分子马达.左边的第一个图是分子结构小电导的力敏通道在大肠杆菌,取自膜蛋白取向(OPM)数据库.
对蛋白质构象变化的全原子模拟可能非常昂贵,这通常需要使用超级计算机。与此同时,以前的许多分析研究都是基于热力学的。例如在机械传导中,马尔金和萨克斯,维金斯和飞利浦,已经开发了热力学模型,将机械敏感通道的门控概率与膜性能联系起来。尽管这些理论模型对力学敏感性的共同特征提供了有趣的见解,但由于模型缺乏结构细节,很难判断它们对任何特定系统的有效性。此外,这些模型中的关键参数通常不是从详细的模拟或实验中获得的。
由于结构细节及其演化往往是力学化学过程的关键正模分析(NMA)对于在原子水平上研究各种分子结构的热力学性质具有很大的吸引力。低频正态模的叠加可以很好地描述生物分子在热扰动或小机械扰动下的结构变化(即集体运动)的整体模式。NMA是基于3N x 3N的对角线海赛矩阵,即质量加权能量矩阵的二阶导数(类似于有限元法中的全局刚度矩阵),其中N为系统中的原子数。然而,由于涉及复杂的经验,获得黑森矩阵可能相当耗时原子势在能量最小化过程中。一个很有前途的解决方法是弹性网络模型(elastic-network model, ENM),由Monique M. Tirion首先提出,这篇论文是本期杂志俱乐部讨论的第一篇论文:
提里奥先生,从单参数原子分析,蛋白质的大振幅弹性运动,物理评论快报,第77卷,第1905页(1996)
在本文中,使用单参数谐波弹簧来模拟相邻原子之间的所有成对相互作用(只要它们在指定的截止距离内)。就低频正态模而言,ENM计算结果与全原子模型计算结果一致。如果只对特征向量感兴趣,那么弹簧常数可以是任意的,唯一相关的参数是截止距离(通常为0.8-1nm)。ENM本身简单,分析性强,计算成本低。此外,模型可以进一步简化,只纳入弹性弹簧网络中的代表性原子(如Cα),被广泛采用。通过给蛋白质的不同组分分配不同的弹簧常数,也可以很容易地进行细化。
为了有效地描述蛋白质的大规模变形,一种基于ENM的简单方法很有意义,这是讨论中的第二篇论文:
w .郑而且美国Doniach,利用简单弹性网络模型对运动蛋白运动的比较研究.美国国家科学院院刊,第100卷,第13253页(2003)
 本文以ENM得到的Hessian矩阵为基础,对某些原子施加位移“边界条件”,通过简单的弹性分析,可以得到ENM中所有原子对应的封闭形式的位移(本文式(6))。为了适应大变形,这种分析可以通过小增量的扰动重复进行。本文重点研究了三个家族的运动蛋白,该方法可以很容易地扩展到其他生物分子。此外,本文的第一作者认为网站,这是一个免费的web服务器,用于计算原子结构的本征模态。
本文以ENM得到的Hessian矩阵为基础,对某些原子施加位移“边界条件”,通过简单的弹性分析,可以得到ENM中所有原子对应的封闭形式的位移(本文式(6))。为了适应大变形,这种分析可以通过小增量的扰动重复进行。本文重点研究了三个家族的运动蛋白,该方法可以很容易地扩展到其他生物分子。此外,本文的第一作者认为网站,这是一个免费的web服务器,用于计算原子结构的本征模态。
一个简单的ENM练习的例子,构象的变化大电导的力敏通道在结核分枝杆菌,在左边的第二个图中给出。在这里,蛋白质受到等双轴张力。使用上述服务器计算黑森矩阵。
DNA的力学特性也很关键,因为在细胞中,DNA会扭曲、弯曲并压缩成一个小体积,从而产生高应力和应变能。事实上,超卷的营养能和应变能之间的交换是控制某些生物生长的一种机制。双螺旋结构的扭转特性对生物序列也很重要。虽然NMA和ENM一般适用于DNA(和RNA),但由于涉及大量的原子,DNA的原子建模仍然相当耗时。由于超高的长截面比(高达10,000,000),DNA可以涂抹出来,就像一根连续的弹性棒,具有特定的弯曲和扭转刚度。杆可以被认为是刚性的,以抵御轴向和横向剪切载荷。如果刚度不受DNA序列和局部环境的影响,在某些情况下,基于经典的Kirchhoff弹性杆模型,可以得到封闭形式的解析解。
由于局部刚度的大小和方向性都强烈依赖于DNA序列,因此杆的刚度应以规定的方式变化,并且还需要将固有曲率和扭曲纳入该方法。此外,DNA的弯曲特性受到其直接离子环境的强烈影响。溶剂分子通过静电相互作用扰动平衡态生物分子的有效能;此外,溶剂分子轰击DNA,从而产生有效的拖曳力,影响动力学。DNA还与蛋白质发生了强而持续的相互作用,蛋白质可以诱导双螺旋轴的特定轨迹,引导弯曲/扭曲/超圈/折叠/环的变形,从而将DNA包装成一个小体积。
虽然很难将所有这些考虑因素纳入一个分析模型,但我们注意到l·马哈而且克劳斯舒尔腾已经迈出了重要的一步,在接下来的论文中
答:Balaeff,库德拉,l·马哈而且k•舒尔腾,使用连续统和统计力学建模DNA环.菲尔。反式。r . Soc。Lond。一个,第362卷,第1355页(2004)
在这篇论文中,他们扩展了经典的Kirchhoff模型,通过蒙特卡罗模拟,加入了序列相关的本征扭曲和曲率、各向异性弯曲刚度、DNA环上的静电相互作用以及溶剂中的过阻尼布朗运动。变形的弹性棒是一个支架,整个DNA的全原子结构可以在其上建立,蛋白质原子和从DNA棒模型中恢复的原子细节之间的相互作用是多尺度方法的基础:
e·维拉,a·巴拉夫,l·马哈而且k•舒尔腾,模拟蛋白质- dna复合物的多尺度方法.多尺度建模。,第2卷,第527页(2004)
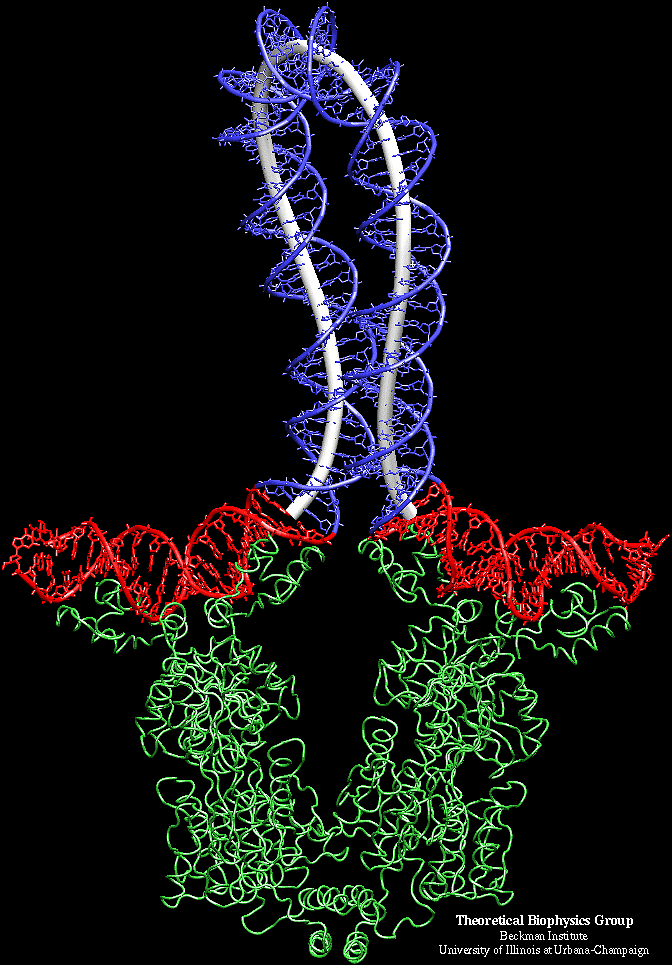 来自紫胶阻遏蛋白-DNA复合物的模型系统(左边第三个图取自Schulten教授的图网站),蛋白质的分子动力学(MD)模拟通过环路开始和结束的“界面”与连续体DNA棒模型耦合。对蛋白质、DNA末端片段和溶剂进行了MD模拟。在MD仿真中,每10,000个时间步后,调用弹性杆模型,更新DNA构型。然后将DNA末端的反作用力代入MD计算区域,并应用于相关原子。这种多尺度方法,用连续的“粗粒”模型处理复合物的某些部分,可能会在其他大分子链中找到重要的应用,以及研究各种生物分子和系统之间的力学化学相互作用。
来自紫胶阻遏蛋白-DNA复合物的模型系统(左边第三个图取自Schulten教授的图网站),蛋白质的分子动力学(MD)模拟通过环路开始和结束的“界面”与连续体DNA棒模型耦合。对蛋白质、DNA末端片段和溶剂进行了MD模拟。在MD仿真中,每10,000个时间步后,调用弹性杆模型,更新DNA构型。然后将DNA末端的反作用力代入MD计算区域,并应用于相关原子。这种多尺度方法,用连续的“粗粒”模型处理复合物的某些部分,可能会在其他大分子链中找到重要的应用,以及研究各种生物分子和系统之间的力学化学相互作用。
上面介绍的蛋白质和DNA的分析模型是经典力学理论的延伸。讨论这些论文的出发点是它们的基本假设,优缺点,以及如何改进和扩展这些模型。请随意发起“头脑风暴”讨论,讨论如何将力学应用于生物分子和系统,同时保持对结构细节的忠实。


评论
温度对DNA、RNA和蛋白质的影响
亲爱的,
我看完了你建议的论文。谢谢你为这个有趣的话题提供了很好的参考。虽然我对生物力学一窍不通,但我还是想说几句。
如何分析温度对DNA、RNA和蛋白质的影响?在先前的分子动力学分析中,DNA、RNA或蛋白质的温度是如何计算的?
如前所述(http://万博manbetx平台m.limpotrade.com/node/1064),温度,以及其他热力学概念,如熵和热力学第二定律,将在分子尺度上变得令人困惑。
挑战在于温度的波动会极大地影响功能DNA, RNA和蛋白质这是一个非常重要的问题,我们不能忽视。
生物分子不能放置在一个孤立的系统中
谢谢红莱。这里讨论的所有组件都必须嵌入在电池及其周围环境中,因此通常假设系统温度为常数。在MD模拟中,这将是将整个系统置于恒温的“水浴”中。热涨落当然会影响系统的平衡和动力学(特别是溶剂分子的平衡和动力学)。Mahadevan和Schulten的著作中对此进行了讨论。
一个细胞里有多少个原子?
西
一个细胞里有多少个原子?按分子计算,这个数是多少?
每个细胞有超过数十亿个分子
Honglai,
实际的数字变化很大,因为单元格的大小可能非常不同。平均来说,一个蛋白质可以包含数百万个分子,而一个细胞很容易包含数十亿个分子。看到在这里粗略估计一下。
所有原子模拟
谢谢你提供的信息。
我现在可以理解,用全原子来模拟蛋白质构象变化的代价是非常昂贵的。
在时间尺度上甚至更昂贵
由于发生在细胞中的典型过程,如机械传导,可能需要几毫秒,如果没有一些偏见的技巧,全原子模拟是根本不可能的。
MD模拟
我认为生物分子动力学过程的最终测试是模拟从随机线圈状态到原生(折叠)状态的折叠过程。几篇论文已经表明,通过MD模拟,可以模拟小于30个氨基酸的小肽的折叠。但是由于大多数酶通常要大得多而且折叠过程在ms的时间尺度上与MD的大系统的ns相比我们还有很长的路要走。
细胞中的时间刻度
细胞生理过程的时间尺度可以超过毫秒,在许多情况下更接近s。在思考生物学和疾病方面,这是另一个巨大的飞跃——在人类中,我们必须尝试并理解细胞中发生的第二类时间尺度的过程与数年出现的疾病之间的关系。在长度尺度上也存在类似的问题:一个细胞有几十微米;一个人(通常!)有几米大。
我对生物力学在分子长度和时间尺度上的进步表示赞赏,但希望这些进步能从更大的角度来看待。要真正利用我们对分子过程的理解来影响医疗保健,在规模上有一个很大的飞跃,这一飞跃与考虑医疗保健至少是研究生物力学的一个很好的理由无关(当然,除了对宇宙和生命意义的基本理解之外!)
你说得对
米歇尔,你说得很对,细胞的过程可能需要更长的时间。我提到了机械传导(这与jClub中讨论的蛋白质通道有更多的力学相关和密切相关)来说明,即使是ms(甚至μs)的过程也很难用全原子模拟来跟踪。就像你说的,长度和时间尺度的挑战几乎存在于所有材料力学问题中,而不仅仅是生物力学。
生命中的时间尺度
我认为你的观点很好,我也经常认为科学在尺度上存在问题。我们只看小事,想要了解大局。有时它是富有成效的,但往往会导致“错误”的结论。但是真的没有其他的方法来做科学,因为如果我们不理解基础,我们就不能理解高级......
这是我的假设。
这是我的假设。
进一步模拟蛋白质结构的还原
也许,把Bahar关于蛋白质建模的论文包括进来会很好。首先,我们可以考虑她在J. Mol. Biol上发表的论文。2003年:Xu, C., Tobi, D.和Bahar, I.,J. Mol.生物学。,333, 153-168 (2003).在她的工作中,证明了从T型(紧张构象:配体-非束缚态)到R型(放松构象:配体-束缚态)的构象转变是由纯弹性力(熵力)诱导的。也就是说,构象跃迁与低频正态模有关,这些正态模负责熵力(热涨落)。为了用NMA来理解构象转变,人们也可以考虑以下论文:
郑伟,布鲁克斯,B.R,Biophys。J。,88, 3109-3117 (2005)
池口,M,上野,J,佐藤,M,木寺,A,理论物理。启。, 078102 (2005)
此外,近年来出现了进一步粗粒化蛋白质结构的问题。在Bahar最近的工作中,大蛋白质的集体运动可以用少量的自由度来表示。具体来说,在她的工作中,GroEL-GroES复合体的结构(~10^4残基)只能用30个节点表示。详情请参阅她的论文:Chennubbhotla, C和Bahar, I。计算机科学课堂讲稿,3909, 379-393 (2006).此外,我还提供了一种粗粒化蛋白质结构的方法。详情请参阅有关文件:严K,白s - c。,安俊华。, Na, S,j .第一版。化学。,在出版社,doi: 10.1002/jcc.20672.
谢谢Kilho
由于期刊俱乐部只能收录3-5篇论文进行讨论,我只选择了这一领域最具代表性(也更具有开拓性)的论文(基于我的浅薄和有限的知识)。我知道你提到的三篇NMA/ENM论文,或多或少都是这个主题的后续。你列出的论文对读者进一步理解这个主题很重要。
但对于粗粒蛋白质,我觉得它更多的是计算而不是分析;另外,与脂质的粗粒化相比,蛋白质的粗粒化还处于初级阶段,仍有许多未解决的问题。此外,我们小组也在这方面进行研究。总的来说,我把这个话题排除在这一期的杂志俱乐部之外,但我欢迎关于这个话题的讨论;或者你可以考虑把它列入期刊俱乐部未来一期的主题。
关于蛋白质粗粒化的未解决问题?
嗨,陈。非常感谢您对我的评论的回复。您说蛋白质粗粒化还处于初级阶段,还有很多问题没有解决。据我猜测,蛋白质的粗颗粒化归因于原生拓扑(由接触映射表示-映射表示原生接触)在蛋白质动力学中发挥作用。也就是说,一般情况下,基于原生接触的蛋白质的刚度矩阵会变成稀疏矩阵,从而导致粗粒化的可能性。我认为这就是为什么从Tirion的粗粒度模型进一步减少模型是可能的。
我有一个简单的问题要问你:你说“有很多没有解决的问题”。你能举例说明吗?据我所知,最具挑战性的问题是了解蛋白质动力学和/或大型蛋白质复合物的力学,我仍在研究中。特别是,我正在利用各种模型约简方法研究大型蛋白质复合体(如GroEL-GroES)的蛋白质动力学。此外,我正在研究蛋白质晶体力学的粗粒度模型。
无论如何,我想听听你关于蛋白质粗粒化的一些未解决的问题的例子,以及你对蛋白质建模的进一步方向的看法。再次感谢您的回复。
更加量化
虽然大多数粗粒度的蛋白质模型可以提供关于运动方向性(如结构转变)的可靠信息,但不能提供关于运动幅度的可靠信息。将已知的结构跃迁与一组正常模态(它们是集合基向量)联系起来通常很简单。基于单一结构来预测结构转变要困难得多;例如,ATP酶与ATP结合后会发生什么。换句话说,你提到的工作很好,但还不能预测。有关更多定量的粗粒度尝试,您可能希望参考格雷格•沃斯的工作。
你打算在生物力学上走多远?
西
那么,你打算在生物力学上走多远?有什么特别的研究方向吗?
生物力学是我们的研究领域之一
Honglai,
我们一般对生物分子的力学感兴趣,但更关注于模拟(而不是本文中的建模)。具体来说,我们正在研究mechanosensitive渠道目前正在开发多尺度仿真协议。这将是我们未来几年的主要方向之一。当然,就像你们一样,我们也同样对能源材料、纳米力学、薄膜、纳米压痕和固流相互作用感兴趣。我正在更新我的部门网页,应该很快发布一些新的信息。